- A+
所属分类:EGFR基因突变
在测序一个新物种时,首先需要对该物种的基因组结构有所了解,包括以下几个内容:
- 基因组大小
- 重复片段大小
- 杂合度大小
基因组越大,杂合度也大,重复片段越大,该物种的组装难度就越大。通常我们会通过genome survery分析,对以上几个指标进行简单评估,核心就是通过kme 分布来进行评估。
对于不同的基因组杂合度,kmer分布如下
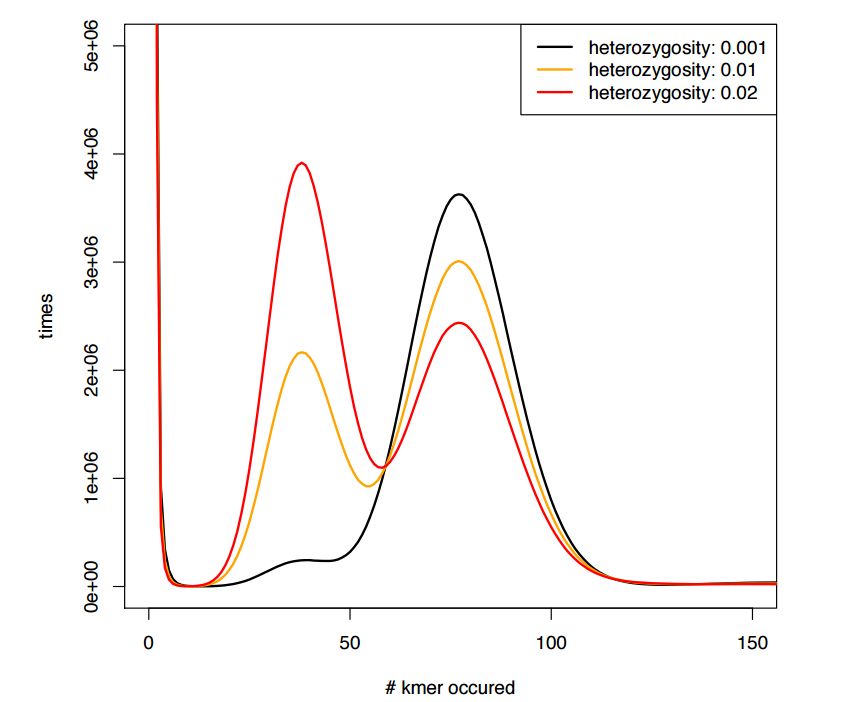
当杂合度为0.001时,只有一个峰;当杂合度为0.01和0.02时,都有多个峰。通过探究杂合度和kmer分布图之间的关系,可以通过kmer分布来评估杂合度。
GenomeScope 软件可以根据kmer分布,评估基因组大小和杂合度,github地址如下
https://github.com/schatzlab/genomescope
安装过程也比较简单,直接下载就可以了
git clone https://github.com/schatzlab/genomescope在软件的安装目录下,genomescopre.R文件是核心的运行脚本,用法如下
Rscript genomescope.R kmer.hist 31 150 test第一个参数 kmer.hist 是jellyfish软件产生的kmer频数分布数据,第二个参数31代表kmer的长度,第三个参数150代表序列读长,第四个参数test 代表输出目录的名称。
在运行过程中,会输出如下信息
GenomeScope analyzing kmer_hist k=31 readlen=150 outdir=test Model converged het:0.0236 kcov:21.7 err:0.00575 model fit:2.21 len:67340147het表示杂合度,为2.36%;len表示基因组大小,为67M左右。输出目录文件列表如下
├── model.txt ├── plot.log.png ├── plot.png ├── progress.txt └── summary.txt通常关注summary.txt,plot.png 这2个文件就可以了。
1. summary.txt
内容如下:
GenomeScope version 1.0 k = 31 property min max Heterozygosity 2.3105% 2.40497% Genome Haploid Length 66,465,582 bp 67,340,147 bp Genome Repeat Length 49,595,265 bp 50,247,848 bp Genome Unique Length 16,870,317 bp 17,092,300 bp Model Fit 80.781% 94.5621% Read Error Rate 0.574733% 0.574733%在该文件中,会给出杂合度,基因组大小。重复片段长度等详细信息。
2. plot.png
示意图如下:
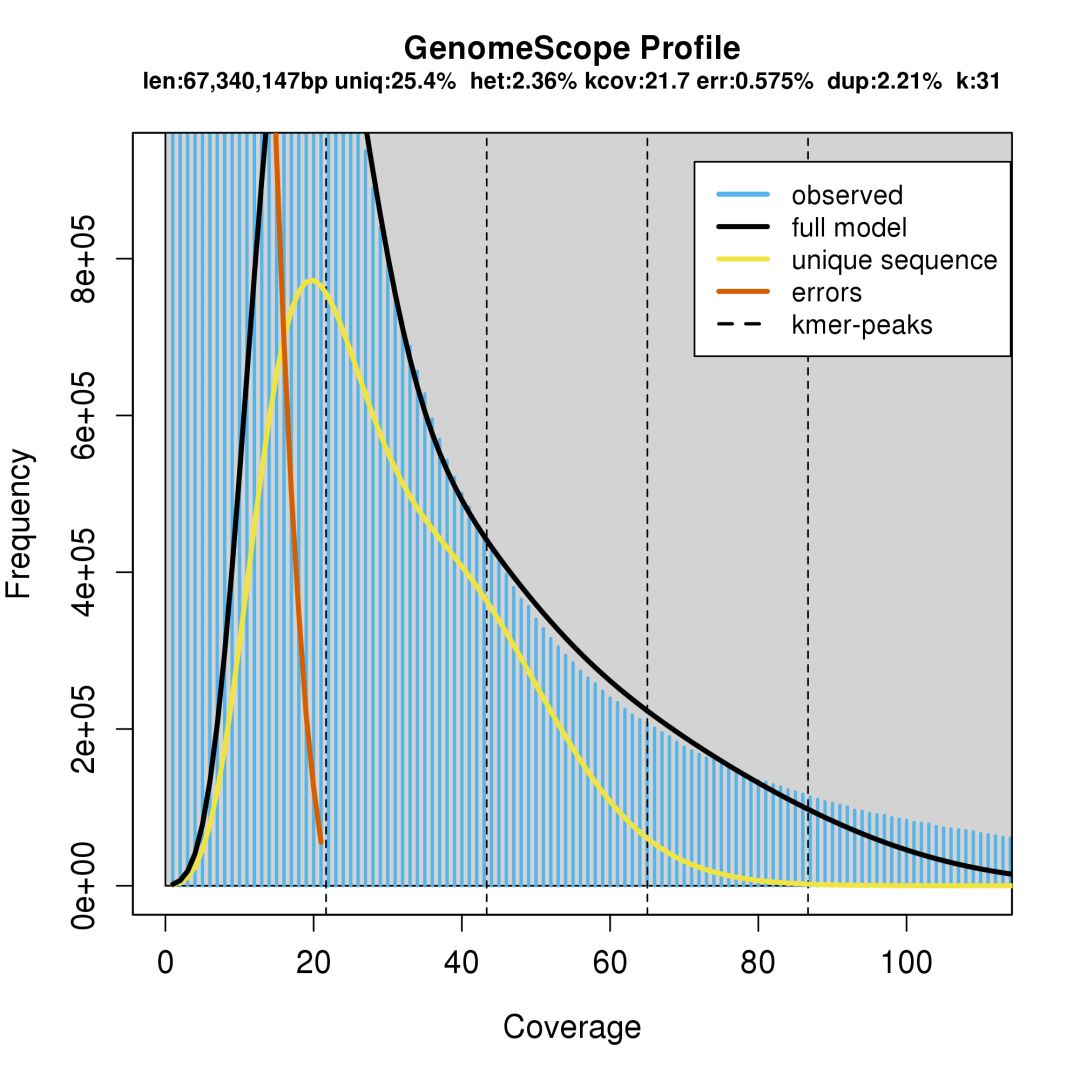
蓝色区域是实际观测到的kmer分布,红色线条下方是一些频数很低的kmer,这些kmer被认为是测序错误,黑色线条下方被认为是可靠的kmer数据,只拿这部分数据来评估基因组的大小,垂直的虚线认为是kmer的几个峰值,黄色线条下方的区域认为是非重复区域的大小。